Abstrakt
Infekcia vírusom SARS-CoV-2 je spojená s prekvapujúcim počtom ochorení. Neobvyklá podobnosť s poruchami krvnej zrážanlivosti a fibrinolytickými poruchami spojenými s amyloidnou chorobou spolu s neurologickými a kardiálnymi problémami nás viedla k skúmaniu amyloidogénnosti proteínu hrotu SARS-CoV-2 (S-proteín). Amyloidné fibrilové testy zmesí peptidových knižníc a teoretické predpovede identifikovali sedem amyloidogénnych sekvencií v rámci S-proteínu. Všetkých sedem izolovaných peptidov vytvorilo počas inkubácie pri 37°C agregáty. Tri 20-aminokyselinové syntetické spike peptidy (sekvencia 192–211, 601–620, 1166–1185) spĺňali tri kritériá amyloidných fibríl: kinetika polymerizácie závislá od nukleácie pomocou ThT, Congo red pozitivita a ultraštrukturálna fibrilárna morfológia. Zložený S-proteín v plnej dĺžke netvoril amyloidné fibrily, ale počas 24 h koinkubácie S-proteínu s proteázou neutrofilnej elastázy (NE) in vitro sa vytvorili fibrily podobné amyloidu so zjavným vetvením. NE účinne štiepila S-proteín, čím došlo k odhaleniu amyloidogénnych segmentov a k akumulácii amyloidogénneho peptidu 194–203, ktorý je súčasťou najviac amyloidogénneho syntetického peptidu hrotu. NE je nadmerne exprimovaný na zapálených miestach vírusovej infekcie. Naše údaje navrhujú molekulárny mechanizmus potenciálnej amyloidogenézy S-proteínu SARS-CoV-2 u ľudí, ktorú uľahčuje endoproteolýza. Perspektíva amyloidogenézy S-proteínu v patogenéze súvisiacej s ochorením COVID-19 môže byť dôležitá pre pochopenie ochorenia a dlhodobého COVID-19.
Koronavírusy využívajú homotrimerný povrchový spike proteín (S-proteín) na prichytenie k ľudským bunkám. Každá podjednotka S-proteínu SARS-CoV-2 obsahuje 1273 aminokyselín. (1) Štyri bežné koronavírusy (OC43, 229E, NL63 a HKU1) infikujú ľudí a kolonizujú dýchacie cesty. Nedávno sa objavili SARS, MERS a od roku 2019 aj SARS-CoV-2, ktoré vedú k závažným ochoreniam. Hoci sú koronavírusové infekcie bežné, až pred COVID-19 bolo zaznamenané také široké rozšírenie komplexných príznakov, ktoré zahŕňajú aj iné orgány ako dýchacie cesty. Patogenéza COVID-19 je multifaktoriálna a komplexná. (2) Závažný COVID-19 zahŕňa syndróm akútnej respiračnej tiesne (ARDS) z vrodených zápalových reakcií imunitného systému, ktoré vedú k poškodeniu pľúc; (3) cytokínovú búrku; (4) poškodenie srdca vrátane zápalu srdcového svalu; poškodenie obličiek; neurologické poškodenie; poškodenie obehového systému, ktoré vedie k zlému prietoku krvi. Medzi dlhodobé príznaky COVID-19 patria pretrvávajúce emocionálne ochorenia a duševné stavy pripomínajúce neurodegeneratívne ochorenia. (2) Čo by mohlo byť základom tejto patogenézy?
Amyloidóza z viacerých "vinných" proteínov sa prejavuje ako systémové a lokalizované poruchy s mnohými fenotypmi, ktoré sa prekrývajú s uvádzanými príznakmi COVID-19. Uvažuje sa, že ťažké zápalové ochorenie vrátane ARDS v kombinácii s agregáciou proteínu SARS-CoV-2 by mohlo indukovať systémovú AA amyloidózu. (5) V súvislosti s neurodegeneráciou sa diskutovalo o neurotropnej kolonizácii a skríženom výseve amyloidných fibríl S-proteínu s cieľom indukovať agregáciu endogénnych proteínov. (6) Pozoruhodné je, že u pacientov s COVID-19 bola zaznamenaná zrážanlivosť krvi spojená s extracelulárnymi agregátmi amyloidových fibríl v krvnom obehu. (7) Hyperkoagulácia/porucha fibrinolýzy sa preukázala v krvnej plazme zdravých darcov experimentálne obohatenej o S-proteín. (7)
Amyloidóza sa spája s mozgovou amyloidovou angiopatiou, poruchou krvnej koagulácie, fibrinolytickou poruchou, (8,9) aktiváciou FXII kallikreínu/kinínu a trombotickým zápalom, (10) čo naznačuje potenciálne súvislosti medzi amyloidogenézou S-proteínu a fenotypmi COVID-19. Predpokladali sme preto potenciálne molekulárne prepojenie medzi S-proteínom a tvorbou amyloidu. Inšpirovaní predchádzajúcimi hypotézami o ľudských a vírusových proteínových amyloidoch a interakciách medzi nimi, (11–13) najmä o spike proteínoch SARS-CoV, (6,14,15) sme si položili otázku: Je S-proteín SARS-CoV-2 amyloidogénny?
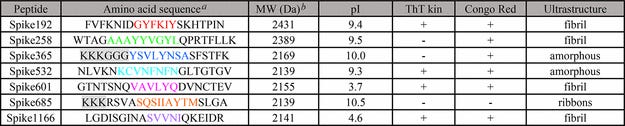
Získali sme knižnicu 316 peptidov (rozdelenú do dvoch podskupín) z peptidového skenovania celého S-proteínu SARS-CoV-2 (ProteinID: P0DTC2) (podporné informácie). V oboch podskupinách peptidov sa in vitro vytvorili amyloidné fibrily (podporné informácie, obrázok S1). Povzbudení týmito výsledkami sme vytvorili 20-AA peptidy z S-proteínu SARS-CoV-2 s plnou dĺžkou. Zamerali sme sa na najviac amyloidogénne sekvencie a na identifikáciu takýchto segmentov sme použili predikčný algoritmus WALTZ (16) (tabuľka 1, podporné informácie).
Tabuľka 1. Sekvencie aminokyselín a vlastnosti syntetických peptidov S-proteínu SARS-CoV-2
a) Farebne priradené rezíduá označujú amyloidogénne segmenty podľa predpovede WALTZ. Šedou farbou sú zvýraznené nenatívne aminokyseliny zavedené kvôli rozpustnosti; b) Teoretická hmotnosť
Identifikovalo sa sedem amyloidogénnych sekvencií rozložených v celom S-proteíne a pomenovaných podľa východiskovej pozície v S-proteíne (obrázok S2, podporné informácie). Všetky predpovedané sekvencie okrem jednej (Spike365) sú v kryoEM štruktúre SARS-CoV-2 Spike v uzavretom stave v konformácii β-sheet. (1) C-koncová časť proteínu (Spike1166) nie je v štruktúre vyriešená.
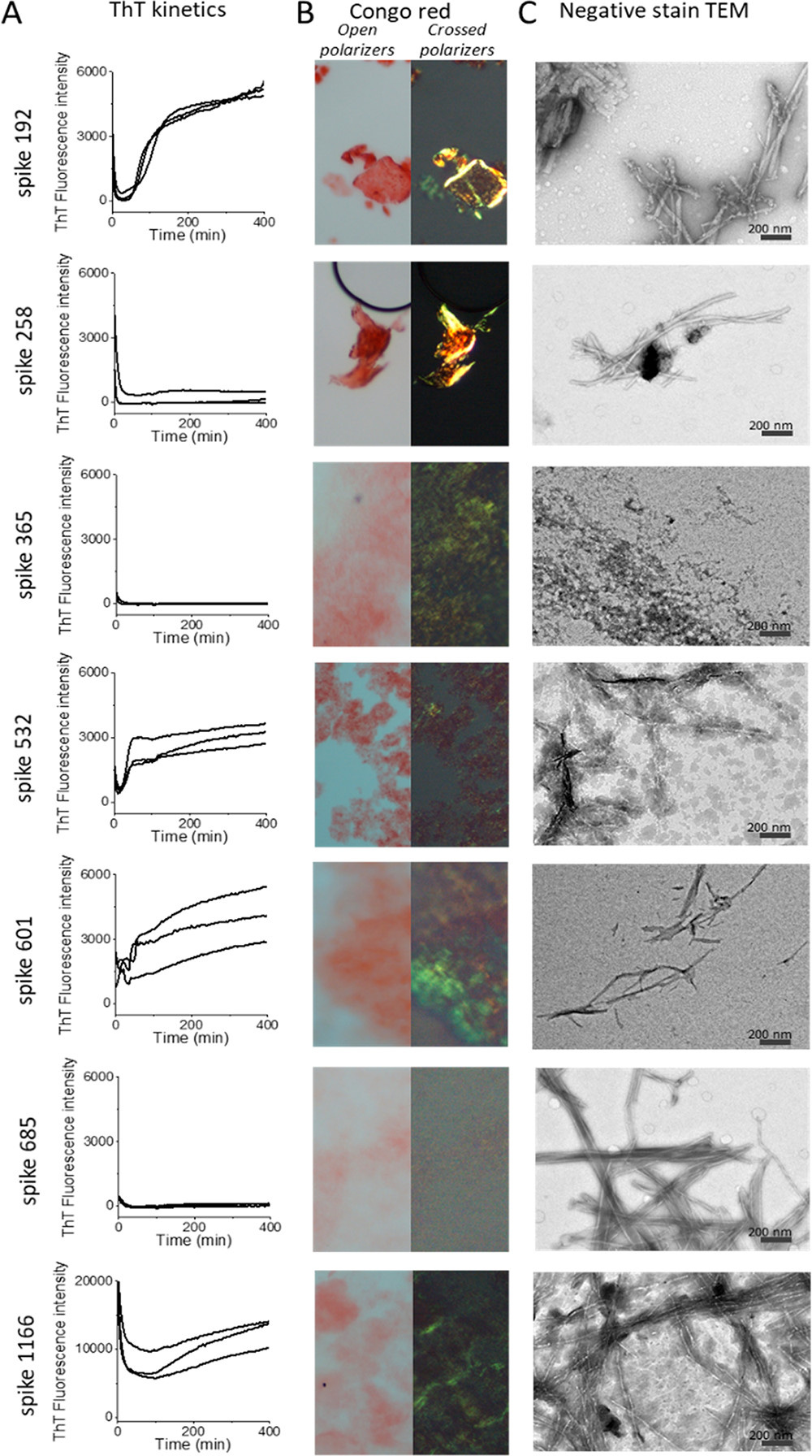
Rozpustené peptidy (0,1 mg/ml, PBS pH 7,5, 10% HFIP) boli sledované z hľadiska kinetiky tvorby amyloidných vlákien in vitro pomocou ThT, dvojlomu konžskej červene (CR) a negatívneho farbenia transmisnej elektrónovej mikroskopie (TEM).
Fibrily z väčšiny syntetických peptidov sa zistili v priebehu niekoľkých hodín aspoň jedným testom (tabuľka 1, obrázok 1). Spike192, Spike601 a Spike1166 spĺňali všetky naše amyloidné kritériá: sigmoidálnu kinetiku ThT, kongofilitu a fibrilárnu ultraštruktúru (obrázok 1, tabuľka 1). Spike192 tvoril výnimočne dobre usporiadané fibrily porovnateľné so zmesou všetkých peptidov (obrázky 1C a S3C).
obr. 1. Testy amyloidných fibríl peptidov SARS-CoV-2 S (0,1 mg/ml). (A) Kinetika tvorby fibríl pri fluorescencii ThT. (B) Mikroskopia s dvojlomom konžskej červenej. (C) Ultraštruktúra TEM s negatívnym farbením.
Aký by bol pravdepodobný mechanizmus tvorby fibríl S-proteínu počas infekcie SARS-CoV-2? S-proteín SARS-CoV-2 je pomerne stabilný (Tm > 50°C) (17) a nebol by ľahko spontánne denaturovaný. Okrem toho sa takýto veľký proteín s komplexným skladaním nebude ľahko nesprávne skladať do amyloidného stavu. Proteolýza je však zrejmým kandidátskym mechanizmom.
Endoproteolýza prekurzorových proteínov je dobre známy molekulárny iniciačný mechanizmus pri viacerých amyloidózach, najmä pri Alzheimerovej chorobe (AβPP), britskej a dánskej demencii (ABri/ADan) a fínskej familiárnej amyloidóze (AGel). Proteolýza proteínov plnej dĺžky je zrejmá aj v mnohých iných amyloidových chorobných ložiskách z ATTR, ALys, AA a ASem1. (18,19)
S-proteín SARS-CoV-2 je počas infekcie proteolyzovaný enzýmami podobnými furínu hostiteľa a serínovými proteázami, ako je transmembránová proteáza, serín 2 (TMPRSS2), na povrchu buniek (20) a ďalej sa proteolyzuje počas zápalu.
Neutrofily sú dominantnou triedou leukocytov a prvou odpoveďou počas akútneho zápalu. Neutrofily sa regrutujú do bronchoalveolárneho priestoru pacientov infikovaných rôznymi respiračnými vírusmi vrátane SARS-CoV-2. (21) Neutrofily pôsobia fagocytózou opsonizovaných patogénov a extracelulárnym uvoľňovaním enzýmov, ako je neutrofilná elastáza (NE). NE je serínová proteáza spojená s obštrukčnými ochoreniami pľúc, ako je cystická fibróza, chronická obštrukčná choroba pľúc (22) a nedostatok alfa-1-antitrypsínu. (23)
Aminokyselinová sekvencia S-proteínu SARS-CoV-2 bola podrobená in silico proteolytickému štiepeniu NE pomocou Expasy Peptide cutter. Jeden z výsledných peptidov, Spike194-213, sa presne zhodoval s peptidom Spike192, len s rámcovým posunom o dve aminokyseliny (podporné informácie), čo naznačuje, že hypotézu možno testovať.
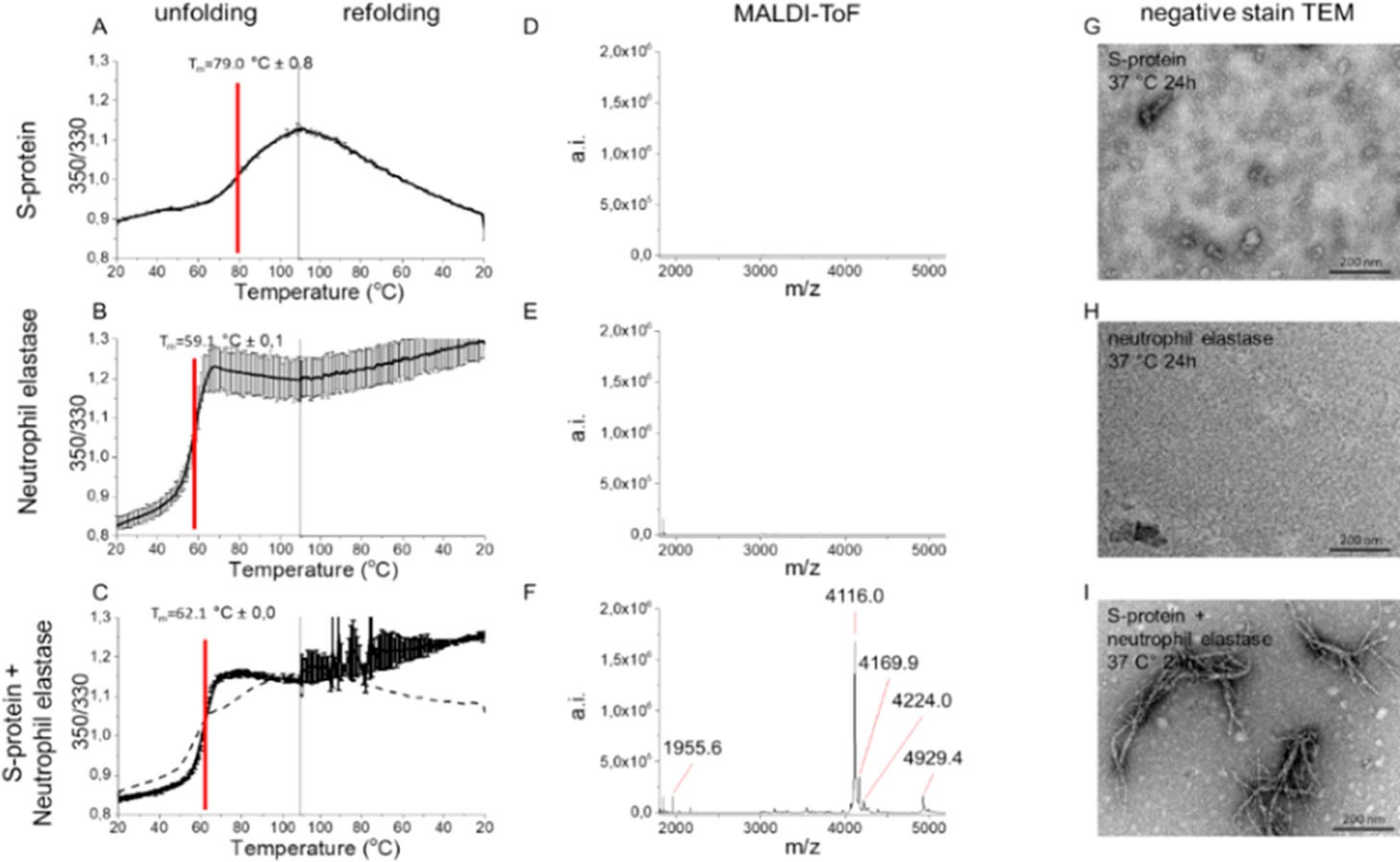
Plnú dĺžku S-proteínu SARS-CoV-2 sme podrobili štiepeniu NE in vitro. S-proteín vykazoval komplexnú trajektóriu tepelného rozkladania s viacerými prechodmi okolo 45–65°C a hlavný prechod rozkladania so stredným bodom denaturácie (Tm) 79°C (obrázky 2A a S4A) s diferenciálnou skenovacou fluorimetriou (DSF) (obrázky 2 a S4), podobne ako literárne hodnoty pre S-proteíny plnej dĺžky, (24) čo potvrdzuje zložený proteín vo východiskovom bode. S-proteín sa po ochladení znovu zložil, aj keď nespolupracoval (obrázok 2A). NE sa zložil ireverzibilne (Tm 59°C) (obrázok 2B). Ko-inkubovaný S-proteín + NE vykazoval zjavný prechod len pre NE a po ochladení sa znovu neskladal, čo naznačuje, že S-proteín bol štiepený NE (obrázok 2C). Hmotnostná spektrometria potvrdila štiepenie, pretože iba experiment S-proteín+NE odhalil peptidové hroty (obrázok 2D–F).
obr. 2. Proteolýza S-proteínu NE vytvára fibrily podobné amyloidu. Termostabilita (A) S-proteínu SARS-CoV-2, (B) NE, (C) S-proteínu+NE meraná pomocou DSF. Prerušovaná čiara v (C) je matematický súčet S-proteínu a NE z (A) a (B), ktorý podporuje štiepenie S-proteínu NE. MALDI-ToF spektrá C18 izolovaných peptidov (D) S-proteínu, (E) NE a (F) S-proteínu+NE (6 h, 37°C). TEM mikrofotografie (G) samotného S-proteínu zobrazujúce očakávané triméry, (H) samotného NE a (I) S-proteínu+NE koinkubovaného pri pH 8.4, 24 h, 37°C
Najdôležitejšie je, že sme objavili tvorbu vlákien podobných amyloidu po proteolytickom štiepení pomocou TEM. Ani NE, ani S-proteín SARS-CoV-2 inkubované samostatne nevytvárali fibrily (obrázok 2G–H). Fibrily sa našli len po ko-inkubácii oboch proteínov (obrázok 2I). Fibrily vykazovali nezvyčajnú morfológiu so zjavným vetvením (obrázok 2I), čo naznačuje účasť proteolyticky zrezaného S-proteínu vo fibrile, čím sa vytvorili uzly pre vetvenie rôznych amyloidogénnych sekvencií (obrázky 2I a S5).
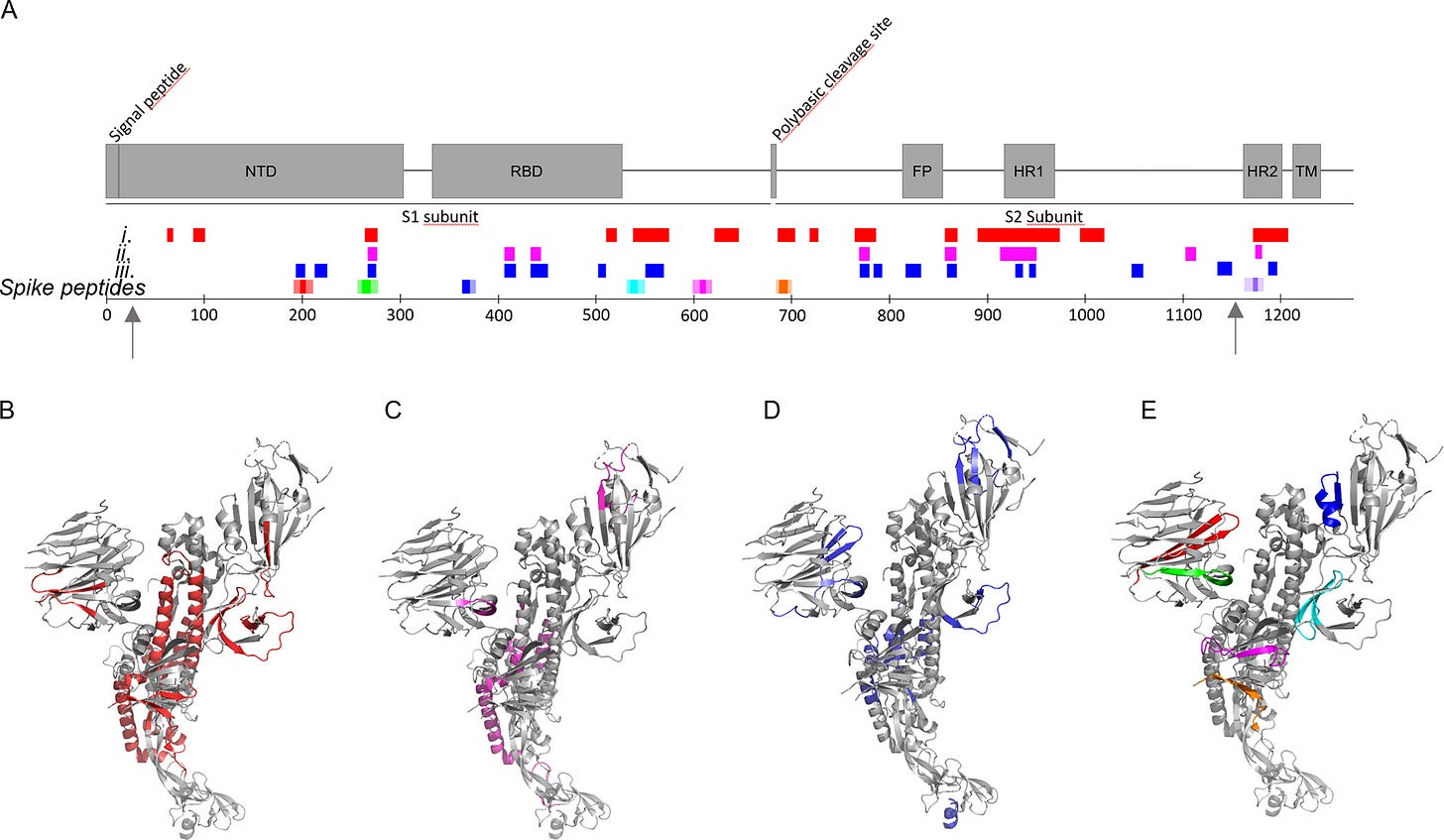
Potom sme vykonali LC-MS/MS analýzu peptidov vytvorených po 1 min. a 6 h trávenia pri prebytku NE nad S-proteínom 2:1. Identifikovali sme 98 peptidov štiepenia NE (tabuľka S1) zo štruktúry S-proteínu a rozdelili sme ich do troch skupín: (i) vytvorené po 1 min. (obrázok 3A, B), (ii) vytvorené po 1 min. a prítomné aj po 6 h (obrázok 3A, C) a (iii) prítomné len po 6 h (obrázok 3A, D). K počiatočnému štiepeniu a ďalšiemu štiepeniu (skupina i) došlo najmä v rámci domény S2 s hojným štiepením domén HR a v C-koncovej časti S1. NTD a RBD boli zasiahnuté oveľa menej (obrázok 3A). Tri pretrvávajúce peptidy vytvorené po počiatočnom štiepení (skupina ii) pochádzali z NTD a RBD. Niekoľko peptidov sa vytvorilo až po 6 h inkubácie (skupina iii). Nápadné je, že peptid zo segmentu 194–203 (FKNIDGYFKI, zahrnutý v Spike192) bol súčasťou tejto skupiny a bol vysoko početný po 6 h (tabuľka S2). Tri peptidy obsahujúce segmenty z našich siedmich hrotových peptidov (obr. 3A, E) sa vytvorili ako voľné peptidy (Spike192, Spike258 a Spike1166), ktoré boli prítomné aj po 6 h koinkubácie, dva sa strávili skoro a zmizli (Spike532 a Spike685) a dva boli pravdepodobne stále prítomné v materskom nakrájanom S-proteíne (Spike365 a Spike601). Preto pozorované vytvorené rozvetvené fibrily (obrázky 2I a S5) pravdepodobne pozostávajú zo zmesi fibríl iniciovaných amyloidogénnym peptidovým semenom, ktoré verbuje poniklovaný S-proteín na predĺženie a rozvetvenie.
obr. 3. Miesta štiepenia NE v rámci S-proteínu plnej dĺžky. Šípky označujú hranice kryoEM štruktúry 27–1146. (A) Peptidy identifikované pomocou LC-MS/MS (tabuľka S1) v korelácii s peptidmi amyloidogénneho hrotu (porovnaj obrázok 1 a tabuľku 1) a štruktúrou domény S-proteínu: po (i) 1 minúte (červená), (ii) 1 minúte a stále pretrvávajúce po 6 hodinách (purpurová), (iii) prítomné len po 6 hodinách inkubácie (modrá). (B–E) Rovnaký farebný kód pre odštiepené peptidové skupiny (i)-(iii) a peptidy s hrotmi mapované na štruktúru protoméru kryoEM S-proteínu PDB 6VXX. (1)
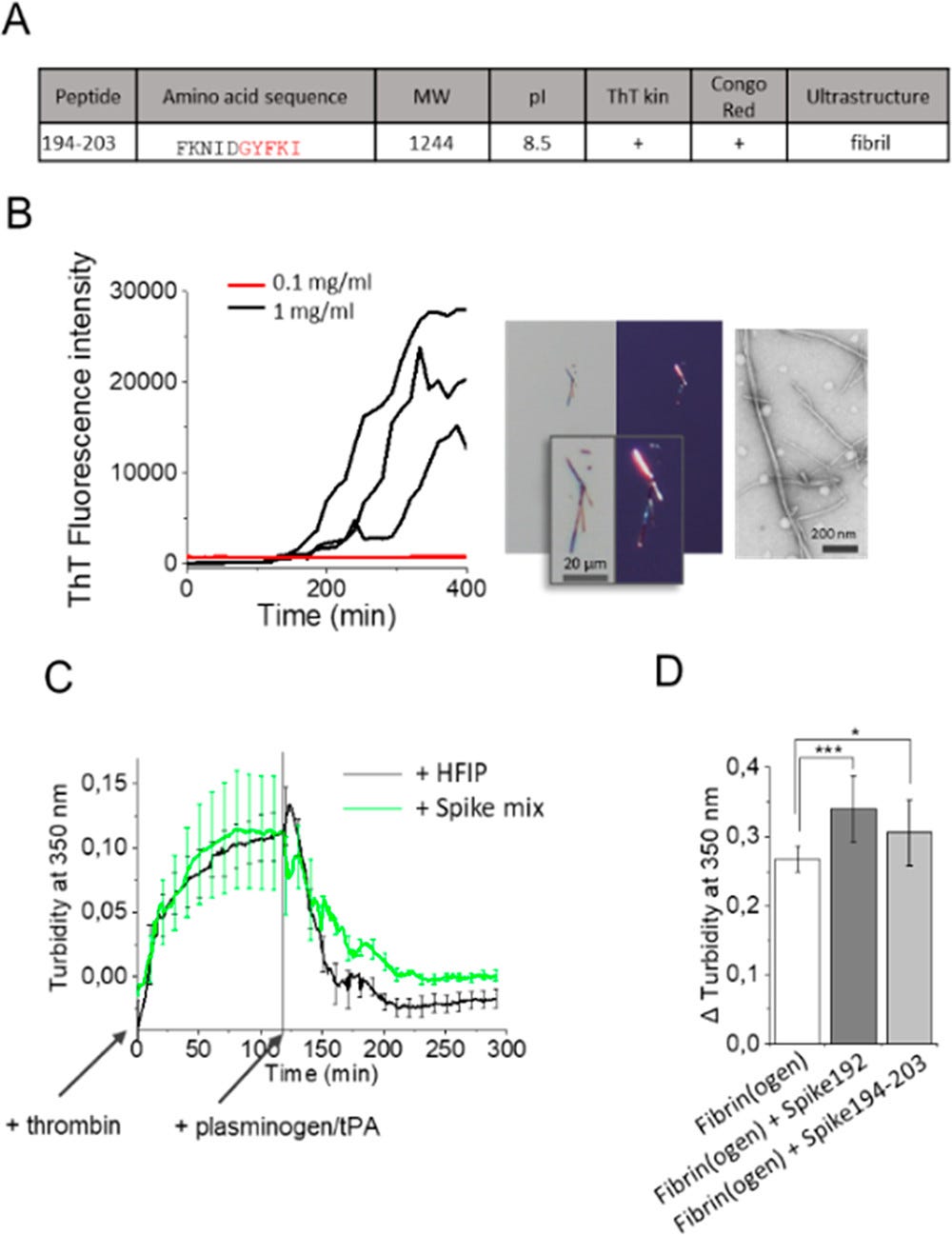
Experimenty s fibriláciou identické s experimentmi pre ostatné špicaté peptidy sme vykonali aj na krátkom fragmente 194–203 (obr. 4A, B). Tento peptid bol menej amyloidogénny ako Spike192 (na vytvorenie fibríl bolo potrebných 1 mg/ml v porovnaní s 0,1 mg/ml pre Spike192) (obrázok 4B). Vytvorené fibrily Spike194-203 však boli amyloidogénne podľa našich troch kritérií, kinetiky ThT, dvojlomu konžskej červenej a ultraštruktúry fibrilárnych vlákien (obrázok 4A, B). Treba poznamenať, že peptidu Spike194–203 chýba jedna aminokyselina v predpokladanej amyloidogénnej sekvencii. Aby sme otestovali význam tohto vynechania aminokyseliny v peptide, vykonali sme jednoduchý in silico mutačný experiment, v ktorom sme nahradili posledný tyrozín v amyloidogénnom segmente Spike192 glycínom, aby sme napodobnili jeho vynechanie. In silico zámena Y204G zrušila predikciu amyloidu (obrázok S6), čo dokazuje, že odstránenie tejto aminokyseliny zmení amyloidogénne vlastnosti peptidu. "Holenie" fibríl je známe pri viacerých amyloidogénnych proteínoch a peptidoch. (25) Je možné, že Y204 sa štiepi po agregácii S-proteínu.
obr. 4. (A) Vlastnosti Spike194–203. (B) Amyloidogénnosť Spike194–203 sledovaná kinetikou ThT, dvojlomom konžskej červenej a TEM. (C) Trombínom aktivovaná tvorba fibrínu a plazminogénom/tPA indukovaná fibrinolýza v neprítomnosti a prítomnosti amyloidných fibríl zmesi siedmich peptidov (tabuľka 1, obrázok S3). (D) Rozdiel v zákale po tvorbe fibrínu a fibrinolýze v neprítomnosti a prítomnosti fibríl Spike192 a Spike194–203 (n = 10–12 opakovaní každej reakcie, korigované na rozptyl pozadia)
Je známe, že S-proteín ovplyvňuje tvorbu pretrvávajúcich mikroskratiek podobných amyloidu v ľudskej krvi, čo je potenciálna patologická príčina dlhých príznakov COVID-19. (26) Vykonali sme trombínom indukovanú premenu fibrinogénu na fibrín s následným testom plazminogénu tPA (27) v prítomnosti a neprítomnosti fibríl peptidu Spike (podporné informácie).
Pridanie 10 μg/ml amyloidných fibríl vytvorených zo zmesi siedmich peptidov Spike (tabuľka 1, obrázok S3) počas tvorby fibrínu znížilo fibrinolýzu (obrázok 4C). Okrem toho pridanie 2% fibríl (zo zásoby 1 mg/ml, celkovo 20 μg/ml) Spike192 a 194–203 zvýšilo perzistentný plazmínom nestráviteľný fibrín (obrázok 4 D). Ako sa očakávalo, amyloidogénnejší Spike192 vyvolal viac fibrínových zrazenín odolných voči plazminu ako Spike194-203. Zdá sa, že náš redukčný test kopíruje výsledky zo vzoriek ľudskej plazmy. (7)
Testovali sme dva fluorescenčné analógy amyloidových značkovačov pozitrónovej emisnej tomografie (PET), CN-PiB (benzotiazolový analóg pittsburskej zlúčeniny B) a DF-9 (stilbénový analóg Florbetabenu), o ktorých je známe, že sa viažu na neurologický amyloid Aβ a srdcový amyloid AL, AA a ATTR, a zistili sme silnú väzbu so súčasnou fluorescenčnou odozvou voči fibrilám Spike192 in vitro (obrázok S7). Ako translačná stratégia môže preto PET zobrazovanie slúžiť ako možnosť pre klinické štúdie na doplnenie tekutej biopsie na hodnotenie amyloidových mikrokvapôčok. (26)
Na záver sme tu navrhli jednoduchý molekulárny mechanizmus, ako môže S-proteín SARS-CoV-2 endoproteolyzovaný NE tvoriť amyloidogénne S-peptidy, ako je segment 194–203, a viesť k odhaleniu viacerých amyloidogénnych segmentov v proteolyticky nakrájanom S-proteíne.
Je možné, že ak tento proces prebieha in vivo, môžu sa na ňom podieľať aj iné amyloidogénne peptidy a S-proteín zrezaný inými proteázami. Zistili sme, že všetky bežné koronavírusy infikujúce ľudí obsahujú amyloidogénne sekvencie (obrázok S8A). Napriek tomu rozsah rozmanitých symptómov COVID-19 nebol predtým zaznamenaný. Segment 194-213 je jedinečný pre SARS-CoV-2 (obrázok S8B), čo by v kombinácii s akútnym zápalom a náborom neutrofilov, o ktorých je známe, že sú častejšie pri COVID-19 v porovnaní s inými vírusovými infekciami, mohlo vysvetliť predpokladanú tvorbu amyloidu spojenú s COVID-19. Treba spomenúť, že amyloidóza je pomerne častá v staršej populácii (18) a jej spojenie s vírusovými infekciami je predmetom diskusie. (13) Nedávne štúdie dokazujú, že pacienti vyliečení z COVID-19 majú zvýšené riziko cukrovky II. typu, ochorenia spojeného s amyloidom. (28,29) Hoci je naša štúdia obmedzená na in vitro zistenia čistých preparátov peptidov a proteínov, výsledky navrhujú zohľadniť amyloidogenézu S-proteínov pri štúdiu COVID-19 a dlhodobých symptómov COVID-19.
preklad: Kishitusumo Takadonomi –> https://tinyurl.com/yxxk3y9a
Ďalšie zaujímavé čítanie nájdete tiež na adresách:
Podporné informácie sú bezplatne k dispozícii na tejto adrese
Skratky
CNS centrálny nervový systém
SARS-CoV-2 koronavírus ťažkého akútneho respiračného syndrómu-2
S-proteín spike proteín
ThT tioflavín T
CN-PiB Kyano-Pittsburská zlúčenina B
TEM transmisná elektrónová mikroskopia
DSF diferenciálna skenovacia fluorimetria
Odkazy
Walls, A. C.; Park, Y. J.; Tortorici, M. A.; Wall, A.; McGuire, A. T.; Veesler, D. Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein. Cell 2020, 181 (2), 281– 292, DOI: 10.1016/j.cell.2020.02.058 [Crossref], [PubMed], [CAS], Google Scholar
Huang, C.; Wang, Y.; Li, X.; Ren, L.; Zhao, J.; Hu, Y.; Zhang, L.; Fan, G.; Xu, J.; Gu, X.; Cheng, Z.; Yu, T.; Xia, J.; Wei, Y.; Wu, W.; Xie, X.; Yin, W.; Li, H.; Liu, M.; Xiao, Y.; Gao, H.; Guo, L.; Xie, J.; Wang, G.; Jiang, R.; Gao, Z.; Jin, Q.; Wang, J.; Cao, B. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. Lancet 2020, 395 (10223), 497– 506, DOI: 10.1016/S0140-6736(20)30183-5 [Crossref], [PubMed], [CAS], Google Scholar
Lipcsey, M.; Persson, B.; Eriksson, O.; Blom, A. M.; Fromell, K.; Hultstrom, M.; Huber-Lang, M.; Ekdahl, K. N.; Frithiof, R.; Nilsson, B. The Outcome of Critically Ill COVID-19 Patients Is Linked to Thromboinflammation Dominated by the Kallikrein/Kinin System. Front Immunol 2021, 12, 627579, DOI: 10.3389/fimmu.2021.627579 [Crossref], [PubMed], [CAS], Google Scholar
Gao, Y. M.; Xu, G.; Wang, B.; Liu, B. C. Cytokine storm syndrome in coronavirus disease 2019: A narrative review. J. Intern Med. 2021, 289 (2), 147– 161, DOI: 10.1111/joim.13144 [Crossref], [PubMed], [CAS], Google Scholar
Sinha, N.; Thakur, A. K. Likelihood of amyloid formation in COVID-19-induced ARDS. Trends Microbiol 2021, 29 (11), 967– 969, DOI: 10.1016/j.tim.2021.03.008 [Crossref], [PubMed], [CAS], Google Scholar
Tavassoly, O.; Safavi, F.; Tavassoly, I. Seeding Brain Protein Aggregation by SARS-CoV-2 as a Possible Long-Term Complication of COVID-19 Infection. ACS Chem. Neurosci. 2020, 11 (22), 3704– 3706, DOI: 10.1021/acschemneuro.0c00676 [ACS Full Text], [CAS], Google Scholar
Grobbelaar, L. M.; Venter, C.; Vlok, M.; Ngoepe, M.; Laubscher, G. J.; Lourens, P. J.; Steenkamp, J.; Kell, Douglas B.; Pretorius, E. SARS-CoV-2 spike protein S1 induces fibrin(ogen) resistant to fibrinolysis: implications for microclot formation in COVID-19. Biosci. Rep. 2021, 41 (8), BSR20210611, DOI: 10.1042/BSR20210611 [Crossref], [PubMed], Google Scholar
Bouma, B.; Maas, C.; Hazenberg, B. P.; Lokhorst, H. M.; Gebbink, M. F. Increased plasmin-alpha2-antiplasmin levels indicate activation of the fibrinolytic system in systemic amyloidoses. J. Thromb Haemost 2007, 5 (6), 1139– 42, DOI: 10.1111/j.1538-7836.2007.02457.x [Crossref], [PubMed], [CAS], Google Scholar
Hammarstrom, P. The bloody path of amyloids and prions. J. Thromb Haemost 2007, 5 (6), 1136– 8, DOI: 10.1111/j.1538-7836.2007.02575.x [Crossref], [PubMed], [CAS], Google Scholar
Maas, C.; Govers-Riemslag, J. W.; Bouma, B.; Schiks, B.; Hazenberg, B. P.; Lokhorst, H. M.; Hammarstrom, P.; ten Cate, H.; de Groot, P. G.; Bouma, B. N.; Gebbink, M. F. Misfolded proteins activate factor XII in humans, leading to kallikrein formation without initiating coagulation. J. Clin. Invest. 2008, 118 (9), 3208– 18, DOI: 10.1172/JCI35424 [Crossref], [PubMed], [CAS], Google Scholar
Munch, J.; Rucker, E.; Standker, L.; Adermann, K.; Goffinet, C.; Schindler, M.; Wildum, S.; Chinnadurai, R.; Rajan, D.; Specht, A.; Gimenez-Gallego, G.; Sanchez, P. C.; Fowler, D. M.; Koulov, A.; Kelly, J. W.; Mothes, W.; Grivel, J. C.; Margolis, L.; Keppler, O. T.; Forssmann, W. G.; Kirchhoff, F. Semen-derived amyloid fibrils drastically enhance HIV infection. Cell 2007, 131 (6), 1059– 71, DOI: 10.1016/j.cell.2007.10.014 [Crossref], [PubMed], [CAS], Google Scholar
Tayeb-Fligelman, E.; Cheng, X.; Tai, C.; Bowler, J. T.; Griner, S.; Sawaya, M. R.; Seidler, P. M.; Jiang, Y. X.; Lu, J.; Rosenberg, G. M.; Salwinski, L.; Abskharon, R.; Zee, C. T.; Hou, K.; Li, Y.; Boyer, D. R.; Murray, K. A.; Falcon, G.; Anderson, D. H.; Cascio, D.; Saelices, L.; Damoiseaux, R.; Guo, F.; Eisenberg, D. S., Inhibition of amyloid formation of the Nucleoprotein of SARS-CoV-2. bioRxiv, March 18, 2021, ver. 1. DOI: 10.1101/2021.03.05.434000v2 (accessed 2022-03-09).[Crossref], Google Scholar
Michiels, E.; Rousseau, F.; Schymkowitz, J. Mechanisms and therapeutic potential of interactions between human amyloids and viruses. Cellular and molecular life sciences: CMLS 2021, 78 (6), 2485– 2501, DOI: 10.1007/s00018-020-03711-8 [Crossref], [PubMed], [CAS], Google Scholar
Zhang, S. M.; Liao, Y.; Neo, T. L.; Lu, Y.; Liu, D. X.; Vahlne, A.; Tam, J. P. Identification and application of self-binding zipper-like sequences in SARS-CoV spike protein. international journal of biochemistry & cell biology 2018, 101, 103– 112, DOI: 10.1016/j.biocel.2018.05.012 [Crossref], [PubMed], [CAS], Google Scholar
Idrees, D.; Kumar, V. SARS-CoV-2 spike protein interactions with amyloidogenic proteins: Potential clues to neurodegeneration. Biochemical and biophysical research communications 2021, 554, 94– 98, DOI: 10.1016/j.bbrc.2021.03.100 [Crossref], [PubMed], [CAS], Google Scholar
Maurer-Stroh, S.; Debulpaep, M.; Kuemmerer, N.; Lopez de la Paz, M.; Martins, I. C.; Reumers, J.; Morris, K. L.; Copland, A.; Serpell, L.; Serrano, L.; Schymkowitz, J. W.; Rousseau, F. Exploring the sequence determinants of amyloid structure using position-specific scoring matrices. Nat. Methods 2010, 7 (3), 237– 42, DOI: 10.1038/nmeth.1432 [Crossref], [PubMed], [CAS], Google Scholar
Upadhyay, V.; Lucas, A.; Panja, S.; Miyauchi, R.; Mallela, K. M. G. Receptor binding, immune escape, and protein stability direct the natural selection of SARS-CoV-2 variants. J. Biol. Chem. 2021, 297 (4), 101208, DOI: 10.1016/j.jbc.2021.101208 [Crossref], [PubMed], [CAS], Google Scholar
Benson, M. D.; Buxbaum, J. N.; Eisenberg, D. S.; Merlini, G.; Saraiva, M. J. M.; Sekijima, Y.; Sipe, J. D.; Westermark, P. Amyloid nomenclature 2020: update and recommendations by the International Society of Amyloidosis (ISA) nomenclature committee. Amyloid 2020, 27 (4), 217– 222, DOI: 10.1080/13506129.2020.1835263 [Crossref], [PubMed], [CAS], Google Scholar
Sipe, J. D. Amyloid proteins: the beta sheet conformation and disease; Wiley-VCH: Weinheim, 2005.[Crossref], Google Scholar
Peacock, T. P.; Goldhill, D. H.; Zhou, J.; Baillon, L.; Frise, R.; Swann, O. C.; Kugathasan, R.; Penn, R.; Brown, J. C.; Sanchez-David, R. Y.; Braga, L.; Williamson, M. K.; Hassard, J. A.; Staller, E.; Hanley, B.; Osborn, M.; Giacca, M.; Davidson, A. D.; Matthews, D. A.; Barclay, W. S. The furin cleavage site in the SARS-CoV-2 spike protein is required for transmission in ferrets. Nature microbiology 2021, 6 (7), 899– 909, DOI: 10.1038/s41564-021-00908-w [Crossref], [PubMed], [CAS], Google Scholar
Johansson, C.; Kirsebom, F. C. M. Neutrophils in respiratory viral infections. Mucosal immunology 2021, 14 (4), 815– 827, DOI: 10.1038/s41385-021-00397-4 [Crossref], [PubMed], [CAS], Google Scholar
Pandey, K. C.; De, S.; Mishra, P. K. Role of Proteases in Chronic Obstructive Pulmonary Disease. Front. Pharmacol. 2017, 8, 512, DOI: 10.3389/fphar.2017.00512 [Crossref], [PubMed], [CAS], Google Scholar
Strnad, P.; McElvaney, N. G.; Lomas, D. A. Alpha1-Antitrypsin Deficiency. N Engl J. Med. 2020, 382 (15), 1443– 1455, DOI: 10.1056/NEJMra1910234 [Crossref], [PubMed], [CAS], Google Scholar
Edwards, R. J.; Mansouri, K.; Stalls, V.; Manne, K.; Watts, B.; Parks, R.; Janowska, K.; Gobeil, S. M. C.; Kopp, M.; Li, D.; Lu, X.; Mu, Z.; Deyton, M.; Oguin, T. H., 3rd; Sprenz, J.; Williams, W.; Saunders, K. O.; Montefiori, D.; Sempowski, G. D.; Henderson, R.; Munir Alam, S.; Haynes, B. F.; Acharya, P. Cold sensitivity of the SARS-CoV-2 spike ectodomain. Nat. Struct Mol. Biol. 2021, 28 (2), 128– 131, DOI: 10.1038/s41594-020-00547-5 [Crossref], [PubMed], [CAS], Google Scholar
Mishra, R.; Sorgjerd, K.; Nystrom, S.; Nordigarden, A.; Yu, Y. C.; Hammarstrom, P. Lysozyme amyloidogenesis is accelerated by specific nicking and fragmentation but decelerated by intact protein binding and conversion. J. Mol. Biol. 2007, 366 (3), 1029– 44, DOI: 10.1016/j.jmb.2006.11.084 [Crossref], [PubMed], [CAS], Google Scholar
Kell, D. B.; Laubscher, G. J.; Pretorius, E. A central role for amyloid fibrin microclots in long COVID/PASC: origins and therapeutic implications. Biochem. J. 2022, 479 (4), 537– 559, DOI: 10.1042/BCJ20220016 [Crossref], [PubMed], [CAS], Google Scholar
Terasawa, F.; Kani, S.; Hongo, M.; Okumura, N. In vitro fibrin clot formation and fibrinolysis using heterozygous plasma fibrinogen from gammaAsn319, Asp320 deletion dysfibrinogen, Otsu I. Thromb Res. 2006, 118 (5), 651– 61, DOI: 10.1016/j.thromres.2005.10.013 [Crossref], [PubMed], [CAS], Google Scholar
Rathmann, W.; Kuss, O.; Kostev, K. Incidence of newly diagnosed diabetes after Covid-19. Diabetologia 2022, 65, 949– 954, DOI: 10.1007/s00125-022-05670-0 [Crossref], [PubMed], [CAS], Google Scholar
Barrett, C. E.; Koyama, A. K.; Alvarez, P.; Chow, W.; Lundeen, E. A.; Perrine, C. G.; Pavkov, M. E.; Rolka, D. B.; Wiltz, J. L.; Bull-Otterson, L.; Gray, S.; Boehmer, T. K.; Gundlapalli, A. V.; Siegel, D. A.; Kompaniyets, L.; Goodman, A. B.; Mahon, B. E.; Tauxe, R. V.; Remley, K.; Saydah, S. Risk for Newly Diagnosed Diabetes > 30 Days After SARS-CoV-2 Infection Among Persons Aged < 18 Years - United States, March 1, 2020-June 28, 2021. MMWR Morb Mortal Wkly Rep 2022, 71 (2), 59– 65, DOI: 10.15585/mmwr.mm7102e2 [Crossref], [PubMed], [CAS], Google Scholar